Title题目
01
Deep learning based label-free virtual staining and classification of human tissues using digital slide scanner
基于深度学习的数字切片扫描仪无标记虚拟染色与人体组织分类
文献速递介绍
02
癌症发病率持续上升,组织病理学仍是诊断金标准,其中H&E染色广泛应用。然而,传统H&E染色存在不可逆、耗时、成本高昂、需专业技术人员等问题,限制了组织后续分析并增加了医疗负担。为解决这些局限性,研究者探索了结合深度学习的无标记光学成像技术进行虚拟染色,以生成诊断相关的H&E等效图像。尽管多种无标记成像模式和深度学习方法(如CycleGAN、CUT)已被开发,但仍面临图像质量、细节保留、单玻片处理效率低下以及难以集成到高通量临床工作流程中的挑战。本文提出了一种基于数字切片扫描仪的深度学习框架,用于无标记虚拟染色和分类,旨在提高诊断图像的解释性、吞吐量和自动化水平。
Aastract摘要
03
苏木精-伊红(H&E)染色是组织病理学的基石,但其不可逆、耗时、需化学品且成本高昂。现有无标记显微镜结合深度学习的方法因成像速度慢、单玻片处理限制等问题,临床应用有限。为解决这些挑战,本文提出了一种高通量虚拟组织学框架,该框架将基于深度学习的虚拟染色和分类与高通量数字多玻片扫描仪相结合。该方法能快速自动化处理来自淋巴结、脑和肝脏等甲醛固定石蜡包埋(FFPE)组织切片的未染色明场(UBF)图像。研究团队采用基于无配对图像到图像转换中对比学习的实例级硬负样本生成(NEGCUT)模型,生成能忠实再现传统染色并保留关键形态特征的虚拟H&E(VHE)图像。此外,他们引入了一个基于残差网络(ResNet)的分类模型,联合利用VHE和UBF图像,实现了95.9%的癌症检测准确率。在盲评中,经董事会认证的病理学家证实VHE图像具有诊断价值,且与传统染色玻片无法区分。该框架利用广泛可用的数字切片扫描仪,提供了一种可扩展且经济高效的传统染色替代方案,实现了高通量组织病理学,并减少了时间、劳动力和化学品使用。
Method方法
04
研究首先制备了人脑、淋巴结、肝脏、甲状腺、骨和结肠组织的FFPE切片,并将其厚度切为4微米。这些未染色切片和经标准H&E染色的切片均使用Pannoramic 250 Flash III、NanoZoomer S360MD和Pannoramic 1000等数字扫描仪以40倍分辨率进行数字化。为确保跨设备兼容性,所有专有格式图像均转换为PNG格式。针对未染色明场(UBF)图像的低对比度问题,进行了对比度增强预处理。图像降采样至10倍放大,并裁剪为256×256像素(虚拟染色)或512×512像素(分类)的瓦片,以10%重叠进行训练和测试。虚拟染色采用NEGCUT模型,该模型通过实例级硬负样本生成进行对比学习,以实现无配对图像到图像的转换,其架构包含编码器-解码器生成器、表示网络和负样本生成器模块。损失函数包括对抗损失、实例级硬负采样对抗对比损失(PatchNCE)和多样性鼓励损失,以确保形态保真度和生成图像的真实感。模型使用Adam优化器训练400个epoch。形态学细节的定量分析通过QuPath软件进行,测量核横截面积、核间距离和每区域细胞核数量。三名病理学家对虚拟H&E(VHE)图像进行盲评,评估核、细胞质、细胞外细节和染色伪影情况。癌症分类采用基于ResNet-18的双模态融合网络,分别处理UBF和VHE图像以提取特征,然后进行拼接并送入全连接层进行二分类。模型使用Adam优化器训练1000个epoch,采用Focal Loss处理类别不平衡,并通过5折交叉验证评估性能。采用Grad-CAM提供分类模型的解释性。
Conclusion结论
05
本研究成功开发了一种基于深度学习的高通量无标记虚拟染色与数字切片扫描图像的自主癌症分类方法。通过NEGCUT虚拟染色模型,未染色明场图像成功转换为具有高度形态相似性的诊断等效虚拟H&E图像,适用于多种组织类型(淋巴结、肝脏、脑、甲状腺和骨骼)。基于ResNet训练的双模态(UBF + VHE)分类模型实现了95.9%的卓越诊断准确率,与真实H&E染色图像的准确率(96.3%)高度一致。病理学家验证进一步证实,虚拟染色图像未损害临床判读所需的重要组织学细节。本方法具有可扩展性和成本效益,有效减轻了传统染色技术的人工劳动、时间和化学品需求,为虚拟染色与临床数字病理学工作流程的快速整合开辟了道路。
Results结果
06
研究成功实现了从正常脑、肝、淋巴结以及转移性淋巴结组织的FFPE样本中高通量获取未染色数字玻片,数字切片扫描仪能在约两小时内自动处理100张玻片,显著提升了吞吐量。NEGCUT模型能生成高质量的VHE图像,通过实例级硬负样本生成结合对比学习,有效保留了精细形态细节。与CycleGAN、CUT和E-CUT等基线模型相比,NEGCUT在FID和KID指标上表现更优(FID 49.69,KID 1.63),视觉上更能清晰描绘细胞核边界、神经纤维网纹理和核仁,且在盲评中被病理学家认为与真实H&E染色图像具有诊断等效性。对淋巴结和肝脏组织的定量形态学分析显示,VHE图像的核面积、核间距和核数量与H&E图像高度一致。模型在甲状腺和骨骼等多样组织类型中也展现出良好的泛化能力。然而,对于结肠等光学对比度极低的组织,虚拟染色效果仍有局限。通过将UBF和VHE图像的特征进行融合,基于ResNet的深度学习模型在癌症检测方面取得了95.89%的准确率和94.84%的F1分数,优于仅使用单一模态(UBF或VHE)的性能,并接近真实H&E图像的分类性能(96.31%准确率)。癌症概率图显示,UBF+VHE模型的预测与H&E模型和病理学家的标注高度一致。Grad-CAM分析表明,模型关注了肿瘤区域的核聚集、不规则轮廓等形态学异常特征,增强了模型的可解释性。
Figure图
07

图1 使用数字切片扫描仪进行基于深度学习的人体组织VHE染色和分类概述。步骤1:从提取的器官中制备福尔马林固定石蜡包埋(FFPE)组织切片。在传统工作流程(红色路径)中,玻片用H&E化学染色。在基于深度学习的工作流程(绿色路径)中,通过数字切片扫描仪获取未染色明场(UBF)图像,并使用NEGCUT模型(用于无配对图像到图像转换中对比学习的实例级硬负样本生成)进行虚拟染色,以生成虚拟H&E(VHE)图像。步骤2:UBF和相应的VHE图像通过双ResNet-18编码器处理,提取的特征被压缩、融合,并使用全连接网络分类为癌性和非癌性类别。该框架提供了传统H&E染色的高通量、无标记替代方案。由BioRender创建。
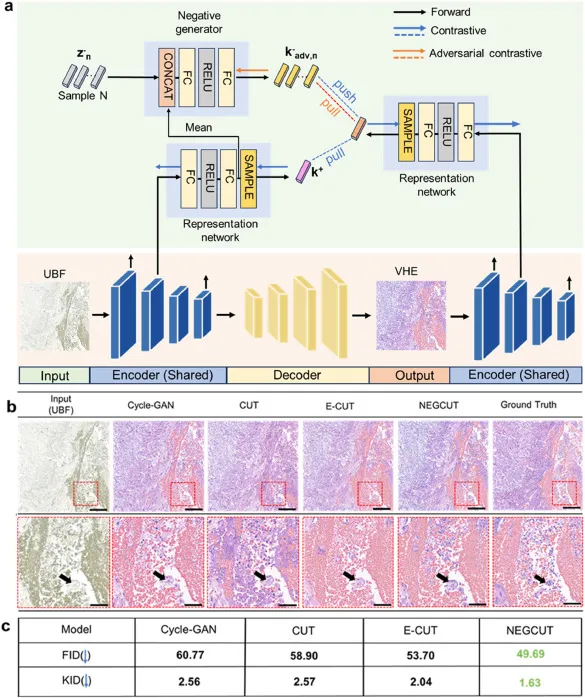
图2 NEGCUT模型整体架构及结果。a NEGCUT模型(修改自Wang等人(Wang et al., 2021))的示意图,用于将未染色明场(UBF)图像转换为虚拟H&E染色(VHE)图像。b CycleGAN、CUT、E-CUT和NEGCUT模型的UBF输入和VHE输出与相应H&E染色图像的视觉比较,使用来自人脑组织的真实样本。比例尺:200微米。每个图像下方显示放大区域,比例尺:50微米。黑色箭头指向代表性细胞核,表示不同网络感知的形态细节差异。c 通过应用Fréchet Inception Distance(FID)和Kernel Inception Distance(KID)指标对图(b)所示模型的虚拟染色性能进行定量评估。较低的FID和KID分数反映了与真实情况更接近的相似性。

图3 人体淋巴结组织切片对比增强未染色明场图像、UBF的VHE图像和H&E染色切片之间的视觉比较。a-c 对比增强未染色明场(CE-UBF)图像;d-f 相应的虚拟H&E染色(VHE)图像,比例尺为1毫米;g-i 相同人体淋巴结组织切片的相应真实组织化学H&E染色图像。a、d和g显示全视野图像,比例尺为1毫米。b、e和h分别显示a、d和g中蓝色高亮区域的放大区域,比例尺为200微米。c、f和i分别显示a、d和g中粉色高亮区域的放大区域,比例尺也为200微米。b、e和h的进一步放大区域分别显示在b1、e1和h1中,着重于详细的细胞形态比较,比例尺为50微米。
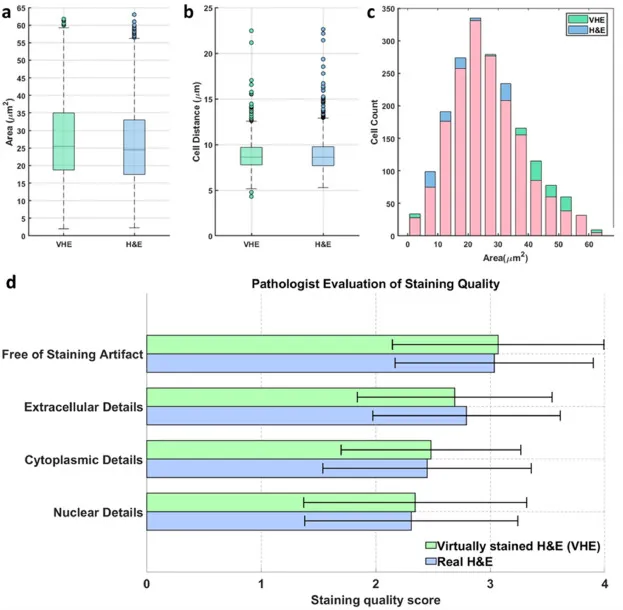
图4 VHE染色图像与H&E图像的定量分析和病理学家评估。a 核横截面积的箱线图显示VHE与真实H&E图像之间具有极好的一致性。b 细胞间距离的箱线图比较显示两种模态下细胞核间距规则。c 核面积分布直方图证实VHE和H&E图像具有相似的细胞大小分布。d 三位经董事会认证的病理学家在四个类别(细胞核细节、细胞质细节、细胞外细节和无染色伪影)上进行盲评,采用4分制(1 = 差,2 = 一般,3 = 好,4 = 优秀)。VHE图像被评定为接近真实H&E,在无染色伪影方面略高。

图5 整体分类网络架构与结果。a 基于ResNet的分类网络完整架构,结合了虚拟H&E染色(VHE)图像和未染色明场(UBF)图像。b 不同输入模态的深度学习(DL)分类结果:单独UBF、单独VHE、建议的UBF和VHE组合方法,以及作为金标准的真实H&E染色图像。c ResNet模型使用UBF和VHE输入生成的癌症概率图(左)与真实H&E输入(中)以及病理学家读取的相应注释(右)的比较。蓝色代表非癌组织,紫色代表癌组织。比例尺:500微米。